
我们提供安全,免费的手游软件下载!

大侠幸会,在下全网同名「算法金」
0 基础转 AI 上岸,多个算法赛 Top
「日更万日,让更多人享受智能乐趣」
t-SNE(t-Distributed Stochastic Neighbor Embedding)是一种用于降维和数据可视化的非线性算法。它被广泛应用于图像处理、文本挖掘和生物信息学等领域,特别擅长处理高维数据。
本文旨在详细介绍 t-SNE 算法的基本概念、数学基础、算法步骤、代码示范及其在不同领域的应用案例。我们还将探讨 t-SNE 的常见误区和注意事项,并与其他降维算法进行对比,以帮助铁子们更好地理解和应用 t-SNE 算法。

by datacamp

t-SNE 是一种非线性降维技术,用于将高维数据映射到低维空间,以便进行可视化。它通过保持高维空间中数据点之间的局部相似性来生成低维空间的表示。这种方法特别适用于揭示复杂数据集中的模式和结构
t-SNE 的核心思想是通过两步过程实现高维到低维的映射。首先,t-SNE 在高维空间中使用高斯分布来计算数据点之间的条件概率。然后,在低维空间中,t-SNE 使用 t 分布来计算相似度,并通过最小化两个分布之间的 Kullback-Leibler 散度(KL 散度)来优化数据点的位置。这个过程可以通过梯度下降法进行优化,从而得到低维空间中的表示。
对看公司很痛苦的同学,可直接跳过公式
在 t-SNE 算法中,首先需要在高维空间中计算数据点之间的相似度。为此,我们使用高斯分布来表示这种相似度。

在低维空间中,t-SNE 使用 t 分布来计算数据点之间的相似度。与高维空间中的条件概率类

为了最小化 KL 散度,t-SNE 使用梯度下降法来优化低维空间中数据点的位置。梯度下降法是一种迭代优化算法,每次迭代更新数据点的位置,使 KL 散度逐渐减小。具体来说,t-SNE 计算 KL 散度对每个数据点位置的梯度,并按照负梯度的方向更新数据点的位置:


在 t-SNE 算法中,首先需要计算高维空间中数据点之间的相似度。具体步骤如下:
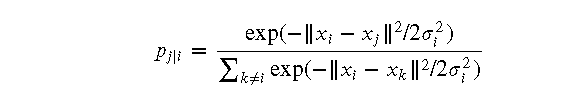
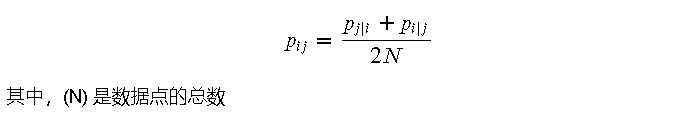
在低维空间中,t-SNE 使用 t 分布来计算数据点之间的相似度。具体步骤如下:
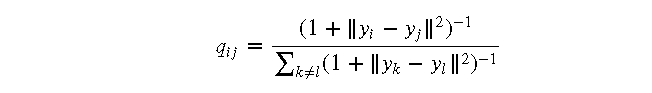
t-SNE 通过最小化高维空间和低维空间之间的相似度分布的 Kullback-Leibler 散度来优化低维空间中数据点的位置。具体步骤如下:
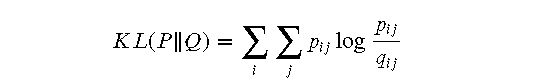
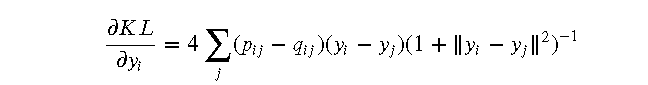
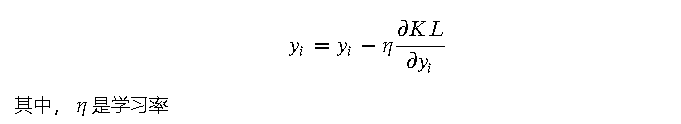
通过上述步骤迭代优化,t-SNE 最终可以得到一个低维空间中的表示,使得高维数据的局部相似性在低维空间中得以保留

在这部分,我们将生成一个带有武侠风格的数据集,包含三个门派的武侠人物。数据集的特征包括武力值、智力值和身法值。我们将使用 t-SNE 进行降维,并展示其可视化效果。接下来,我们会调整 t-SNE 的参数以观察其对降维结果的影响。
import numpy as np
import pandas as pd
import matplotlib.pyplot as plt
from sklearn.manifold import TSNE
from sklearn.datasets import load_iris
# 生成武侠人物数据集
np.random.seed(42)
num_samples_per_class = 50
data = {
'武力值': np.hstack([np.random.normal(loc=5, scale=1, size=num_samples_per_class),
np.random.normal(loc=7, scale=1, size=num_samples_per_class),
np.random.normal(loc=9, scale=1, size=num_samples_per_class)]),
'智力值': np.hstack([np.random.normal(loc=3, scale=1, size=num_samples_per_class),
np.random.normal(loc=5, scale=1, size=num_samples_per_class),
np.random.normal(loc=7, scale=1, size=num_samples_per_class)]),
'身法值': np.hstack([np.random.normal(loc=1, scale=1, size=num_samples_per_class),
np.random.normal(loc=2, scale=1, size=num_samples_per_class),
np.random.normal(loc=3, scale=1, size=num_samples_per_class)]),
'门派': np.hstack([np.full(num_samples_per_class, '少林'),
np.full(num_samples_per_class, '武当'),
np.full(num_samples_per_class, '峨眉')])
}
df = pd.DataFrame(data)
# 将类别标签转换为数字
df['门派'] = df['门派'].astype('category').cat.codes
# 打印前几行数据
print(df.head())
# t-SNE 降维
X = df[['武力值', '智力值', '身法值']]
y = df['门派']
tsne = TSNE(n_components=2, random_state=42)
X_tsne = tsne.fit_transform(X)
# 可视化 t-SNE 结果
plt.figure(figsize=(10, 7))
scatter = plt.scatter(X_tsne[:, 0], X_tsne[:, 1], c=y, cmap='viridis')
plt.colorbar(scatter, ticks=[0, 1, 2], label='门派')
plt.clim(-0.5, 2.5)
plt.title('t-SNE 结果可视化')
plt.xlabel('t-SNE 维度 1')
plt.ylabel('t-SNE 维度 2')
plt.show()
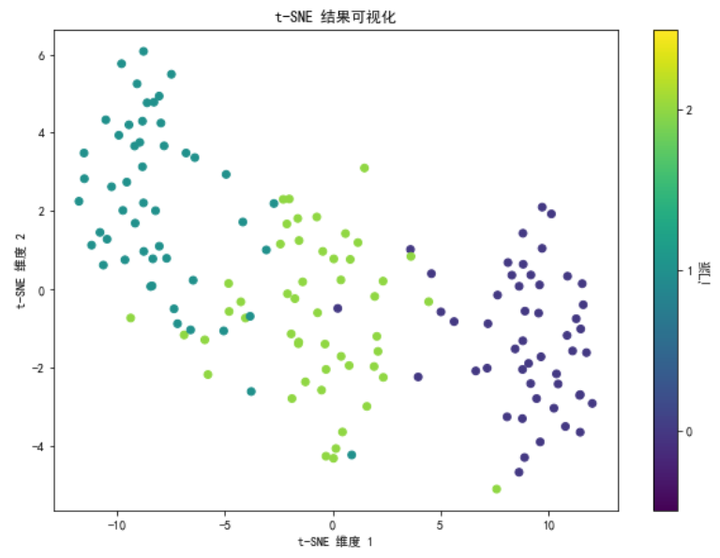
解释与结果解读
在基本实现中,我们生成了一个包含武侠人物特征和门派标签的数据集。数据集中的武侠人物分别来自少林、武当和峨眉三个门派。我们使用 t-SNE 将数据降维到二维,并可视化其结果。不同颜色表示不同的门派,从图中可以看到,同一门派的武侠人物在降维后的二维空间中聚集在一起,而不同门派的武侠人物则分布在不同的区域。
接下来,我们调整 t-SNE 的两个关键参数:perplexity 和 learning_rate,并观察它们对降维结果的影响。
# 调整 perplexity 参数
tsne_perplexity = TSNE(n_components=2, perplexity=30, random_state=42)
X_tsne_perplexity = tsne_perplexity.fit_transform(X)
plt.figure(figsize=(10, 7))
scatter_perplexity = plt.scatter(X_tsne_perplexity[:, 0], X_tsne_perplexity[:, 1], c=y, cmap='viridis')
plt.colorbar(scatter_perplexity, ticks=[0, 1, 2], label='门派')
plt.clim(-0.5, 2.5)
plt.title('t-SNE 结果 (perplexity=30)')
plt.xlabel('t-SNE 维度 1')
plt.ylabel('t-SNE 维度 2')
plt.show()
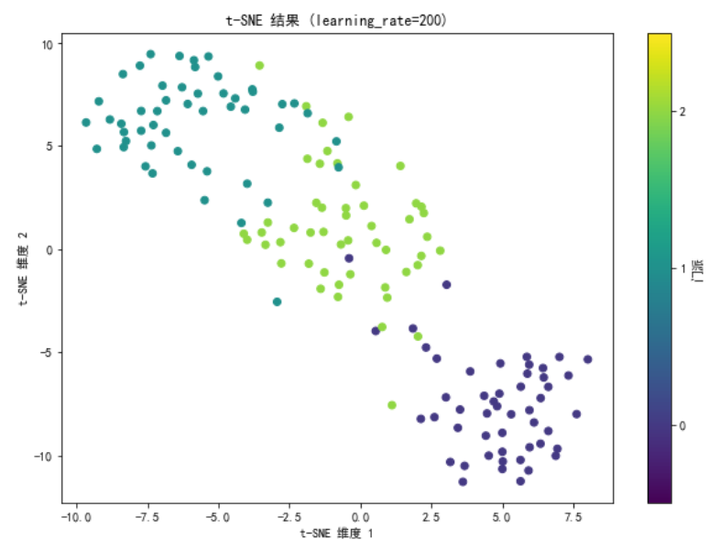
# 调整 learning_rate 参数
tsne_learning_rate = TSNE(n_components=2, learning_rate=200, random_state=42)
X_tsne_learning_rate = tsne_learning_rate.fit_transform(X)
plt.figure(figsize=(10, 7))
scatter_learning_rate = plt.scatter(X_tsne_learning_rate[:, 0], X_tsne_learning_rate[:, 1], c=y, cmap='viridis')
plt.colorbar(scatter_learning_rate, ticks=[0, 1, 2], label='门派')
plt.clim(-0.5, 2.5)
plt.title('t-SNE 结果 (learning_rate=200)')
plt.xlabel('t-SNE 维度 1')
plt.ylabel('t-SNE 维度 2')
plt.show()
解释与结果解读
通过这些示例和详细解释,可以更好地理解 t-SNE 算法及其在实际数据集中的应用效果。希望这能帮助你更好地掌握 t-SNE 的使用方法和参数调优技巧。
每天一个简洁明了的小案例,如果你对这类文章感兴趣,
欢迎订阅、点赞和分享哦~
t-SNE 在图像数据降维中非常有效。以下示例展示了如何将 t-SNE 应用于图像数据降维和可视化。我们将使用手写数字数据集(MNIST)进行演示。
import numpy as np
import pandas as pd
from sklearn.manifold import TSNE
from sklearn.datasets import fetch_openml
from sklearn.preprocessing import StandardScaler
from sklearn.decomposition import PCA
import matplotlib.pyplot as plt
# 加载 MNIST 数据集
mnist = fetch_openml('mnist_784', version=1)
X = mnist.data / 255.0 # 将数据归一化到 [0, 1] 区间
y = mnist.target
# 随机选择 10000 个数据点
np.random.seed(42)
indices = np.random.choice(X.shape[0], 10000, replace=False)
X_subset = X.iloc[indices]
y_subset = y.iloc[indices]
# 标准化数据
scaler = StandardScaler()
X_scaled = scaler.fit_transform(X_subset)
# 应用 t-SNE 进行降维
tsne = TSNE(n_components=2, perplexity=30, n_iter=1000, random_state=42)
X_tsne = tsne.fit_transform(X_scaled)
# t-SNE 可视化结果
plt.figure(figsize=(12, 8))
scatter_tsne = plt.scatter(X_tsne[:, 0], X_tsne[:, 1], c=y_subset.astype(int), cmap='tab10', s=1)
plt.legend(*scatter_tsne.legend_elements(), title="Digits")
plt.title('MNIST 数据集的 t-SNE 可视化')
plt.xlabel('t-SNE 维度 1')
plt.ylabel('t-SNE 维度 2')
plt.show()
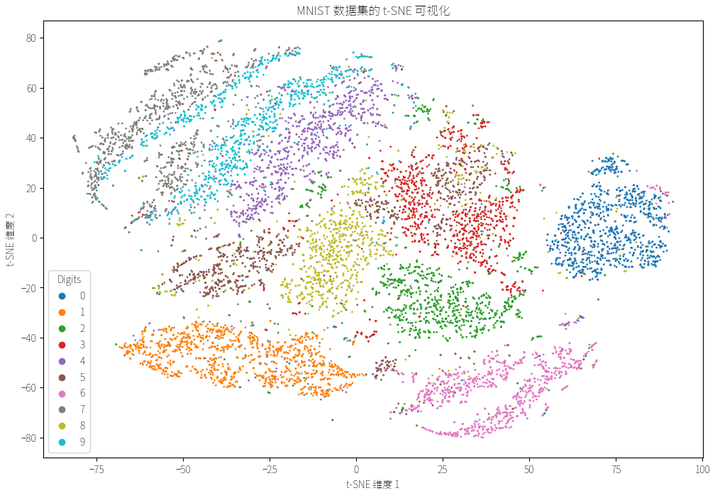
t-SNE 也广泛应用于文本数据降维。以下示例展示了如何将 t-SNE 应用于文本数据降维和可视化。我们将使用新闻组数据集进行演示。
在生物信息学中,t-SNE 常用于基因表达数据的降维和可视化。以下示例展示了如何将 t-SNE 应用于单细胞 RNA 序列数据的降维和可视化。

t-SNE 的计算复杂度较高,对于大规模数据集,计算时间和内存消耗都非常大。因此,t-SNE 不适合直接应用于大数据集。在处理大数据集时,可以考虑以下几种方法:
t-SNE 的降维效果对参数的选择非常敏感。两个关键参数是 perplexity 和 learning_rate。perplexity 参数控制每个数据点的有效邻居数量,一般设置在 5 到 50 之间;learning_rate 参数控制梯度下降的步长,通常设置在 10 到 1000 之间。以下是一些经验性的参数选择建议:
t-SNE 的可视化结果虽然直观,但有时会产生误导。需要注意以下几点:

PCA(主成分分析)和 t-SNE 是两种常用的降维算法,但它们的原理和应用场景有所不同:
LLE(局部线性嵌入)和 t-SNE 都是非线性降维方法,但它们的实现方式不同:
每种降维算法都有其优劣势,选择合适的算法取决于具体的数据集和任务需求:
通过以上对比,可以更好地理解不同降维算法的适用场景和特点,从而选择最适合具体任务的算法
降维和聚类是数据分析中的两种不同但相关的方法:
虽然降维和聚类有不同的目标,但它们可以结合使用。例如,降维可以用于将高维数据投影到低维空间,从而便于进行聚类分析
UMAP(Uniform Manifold Approximation and Projection)是另一种非线性降维方法,常用于与 t-SNE 进行比较:
MDS(多维尺度分析)和 t-SNE 都是用于降维和数据可视化的算法:
通过以上的详细介绍,希望大侠对 t-SNE 算法有了更深入的理解和认识。在实际应用中,结合数据特点和任务需求,选择合适的降维方法,才能发挥数据分析和可视化的最大效用。
- 科研为国分忧,创新与民造福 -

日更时间紧任务急,难免有疏漏之处,还请大侠海涵
内容仅供学习交流之用,部分素材来自网络,侵联删
如果觉得内容有价值,烦请大侠多多 分享、在看、点赞,助力算法金又猛又持久、很黄很 BL 的日更下去;
同时邀请大侠 关注、星标 算法金,围观日更万日,助你功力大增、笑傲江湖
热门资讯